超洁净分子酶——核酸残留的清除秘籍!
来源:瀚海生物市场中心 时间:2024-11-13 作者:瀚海新酶 浏览量:51
诊断迷雾:核酸残留引发的真假信号
在分子生物学研究和诊断应用中,病原体呈现多样化和复杂化的发展趋势,多数患者存在病原体不明的困境,如何快速并准确地检测出病原体类型面临着严峻的挑战,其中背景菌核酸残留已经成为影响检测结果准确性和可靠性的关键因素之一。
采用 PCR、tNGS 和 mNGS 等技术检测呼吸道病毒、肠道菌群、血流感染等病原体时,污染的背景菌核酸可能会淹没低丰度的靶标核酸,或者与靶标核酸一起被检出,从而影响靶标检出灵敏度或造成假阳性结果[1,2],给医生的诊断和用药造成困扰;此外,在诊断试剂开发过程中,由于市场上的分子酶原料的质量良莠不齐,核酸残留过高会影响扩增产物的特异性和准确性[3],进而影响实验的结果和分析,给开发者带来极大的阻碍和挑战。
经过实践发现,分子诊断关键酶原料只有在核酸残留极低(如 Taq DNA 聚合酶 HCD ≤ 0.001copies/U)甚至达到无检出的水平才能满足日益提升的体外诊断试剂的性能需求。
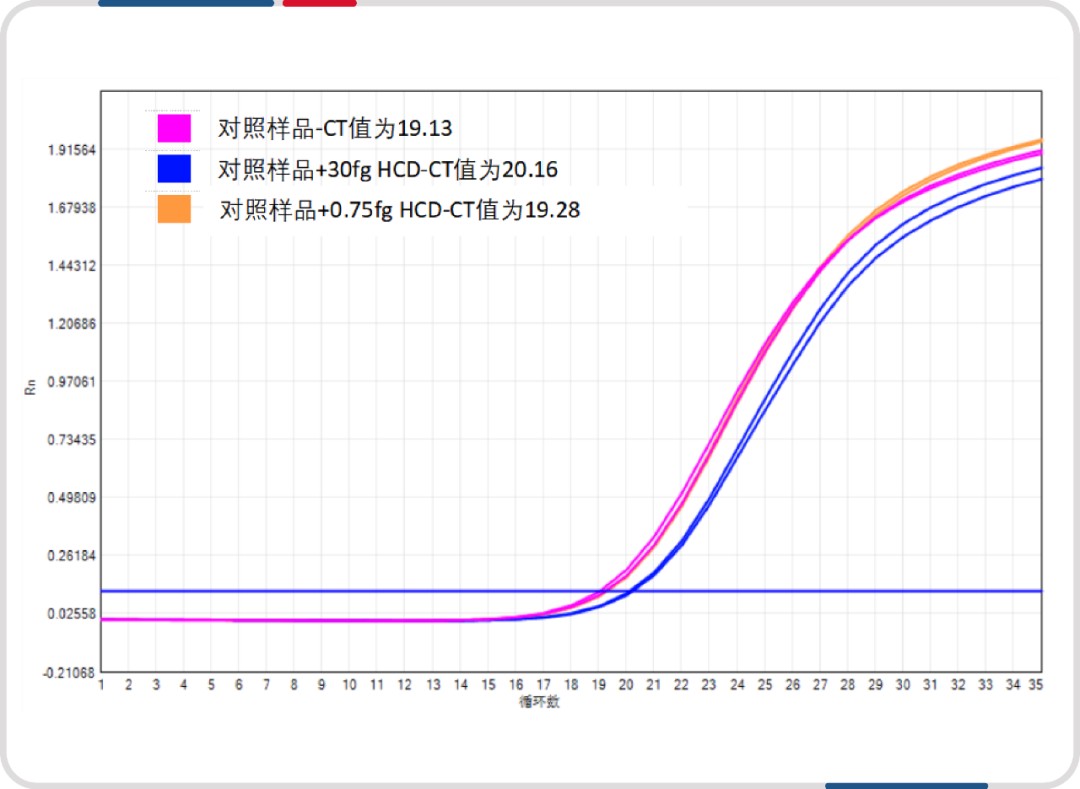
图注:核酸残留较多易产生非特异性扩增,使扩增效率降低,CT 值推后,从而影响检测的灵敏度
02 净化之道:高洁净分子酶开发指南
然而想要彻底清除核酸残留并非易事,一方面宿主核酸分子极其稳定,由于其特殊的荷电性能还极易与带正电荷的蛋白结合直至进入终产品;另一方面在生产过程中,待测样本、试剂、设备、环境以及操作者等也可能会引入核酸污染,因此想要彻底清除核酸残留,既要控制外源性污染,又要做好内源性杂质去除,丰富的技术手段和严格的质量控制结合,才能实现超净分子酶的开发目标。
外围防线:外源性核酸残留的清除行动
可能引入外源核酸污染的环节有很多,在研发和生产过程中,合理规划实验室或生产空间至关重要,应将不同的操作区域严格区分,以减少交叉污染的机会。
定期使用适合的消毒剂(如 70%-80% 乙醇溶液、次氯酸钠溶液等)对台面和仪器表面进行清洁有助于环境核酸的控制,研究表明次氯酸钠对去除气溶胶尤为有效[5]。生产操作时建议按照 GMP 的质量管理体系进行,应穿戴干净的手套、口罩和实验服等,减少人为污染,避免用手直接接触实验材料,尽可能使用移液器等工具代替手动操作,使用封闭式系统进行分子酶的制备。
值得一提的是,在实际测试中瀚海新酶研发部发现物料甘油和枪头的核酸残留水平存在较大差异,耗材端的污染引入可能会对终产品的质量带来致命的打击。尽可能使用一次性耗材,避免重复操作,能极大程度地减少外源污染的引入。
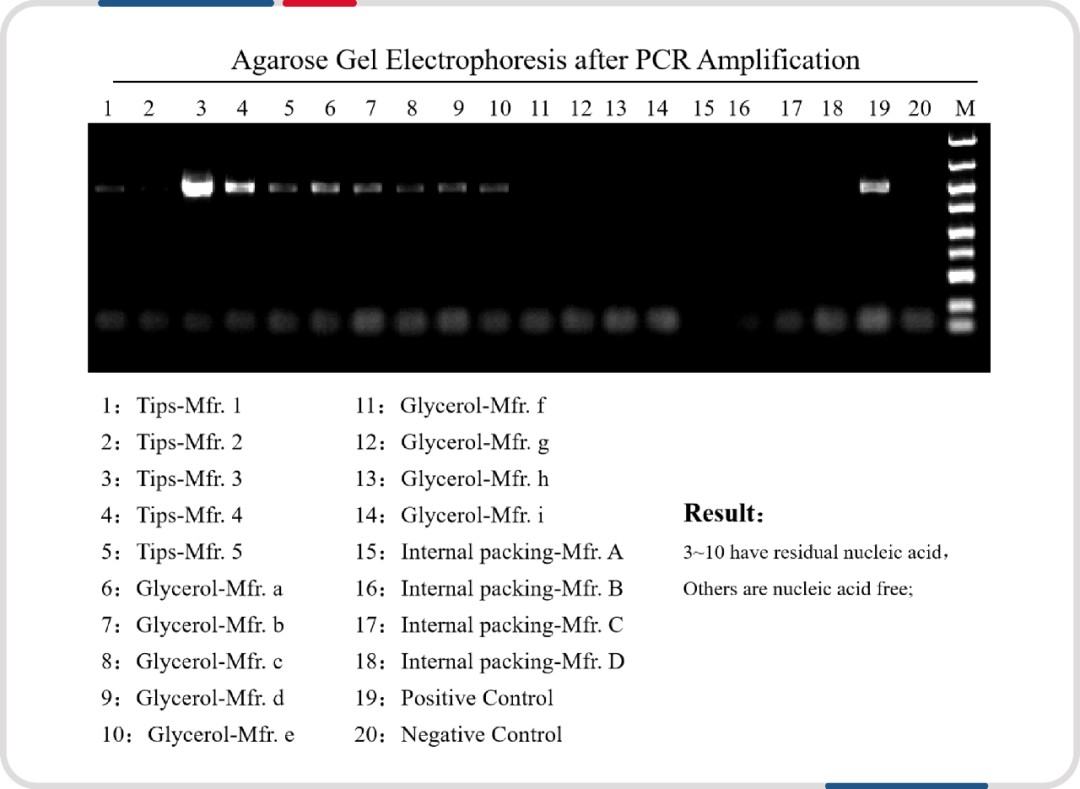
图注:不同厂家的吸头、甘油和内包装的核酸残留具有较大的差异
内部净化:内源性核酸残留的去除策略
良好的工艺往往在样品前处理阶段能够去除大量的核酸污染,再通过后续的层析步骤进一步去除,以保证产品能稳定达到低核酸残留的水平。
许多层析介质已经充分被证实对核酸的去除有明显效果;除此之外,合适的层析条件(如 pH 值、样品装载量和目标峰的收集方式等)也同样重要;最后需要对不同类型的层析进行组合优化来达到包含收率、纯度在内的整体工艺目标。
1) 前端核酸初步净化对于细胞裂解后的样品,常常采用沉淀法进行核酸的大量去除,利用核酸分子带有大量的负电荷,使用带有正电荷的物质与其形成电荷中和,进而形成沉淀,例如聚乙烯亚胺(PEI)[6]、硫酸链霉素、鱼精蛋白沉淀等试剂。
此外,通过酶消化的方式也能去除核酸的污染[7],Karina 等[8] 人采用 Benzonase和
Denarase 对 DNA 和腺病毒的水解时间和剂量的优化,可以看出酶解效果受底物影响。在使用瀚海新酶的 Super UltraNuclease(HBP000102)或 DNase I(HBP000910)等酶类试剂时,需要对剂量、温度和时间进行 DOE 分析,确认最佳条件。
核酸酶和沉淀剂的组合使用(纯化伴侣,HBP000101),更有效解决长链核酸导致的黏性问题,同时还能提高样品的澄清度。
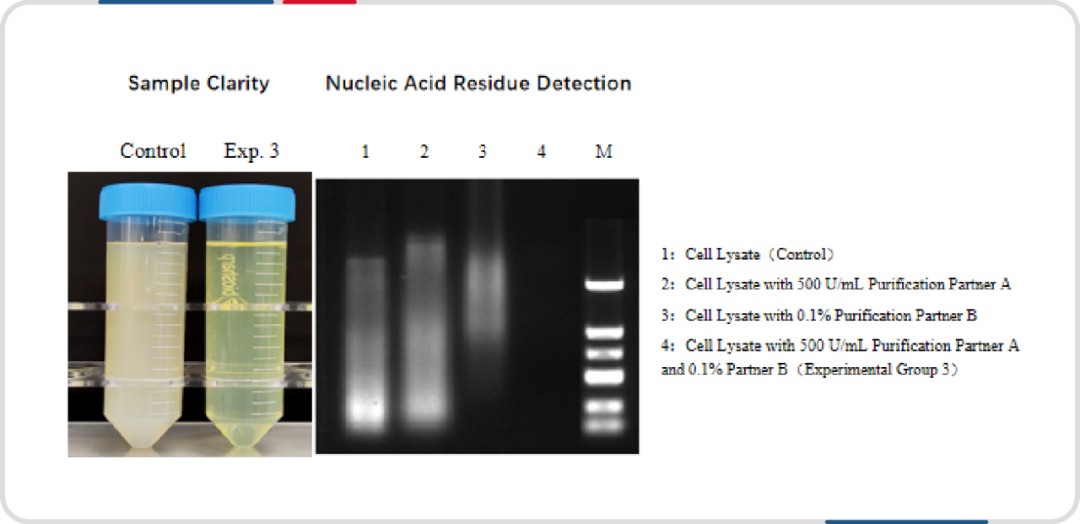
图注:纯化伴侣的使用能让裂解液更加澄清 (实验组 3),同时去除大量的核酸残留(泳道 4)
2)层析多重净化,核酸无处遁形核酸具有较强的负电荷特点,含有亲水性的碱基,整体表现出较弱的疏水性,与常规蛋白类的生物分子有显著的差异。因此,肝素层析、阴离子交换层析、凝胶层析、疏水层析及复合层析较多地用于生物制品中核酸的去除。
→肝素层析
肝素的结构中存在大量的磺酸基与羧基,富含阴离子的结构在一定程度上模拟了链状的核酸,使得肝素具备与 DNA 结合蛋白互作的功能。由血浆制备的多个生物制品如 Protein C、纤维蛋白原等分离过程中都常用到肝素层析,以保证核酸残留符合标准[9]。
通过肝素层析的初步精纯,大肠表达的重组尿嘧啶-DNA 糖基化酶的宿主核酸残留(HCD)可降低约 20 倍。
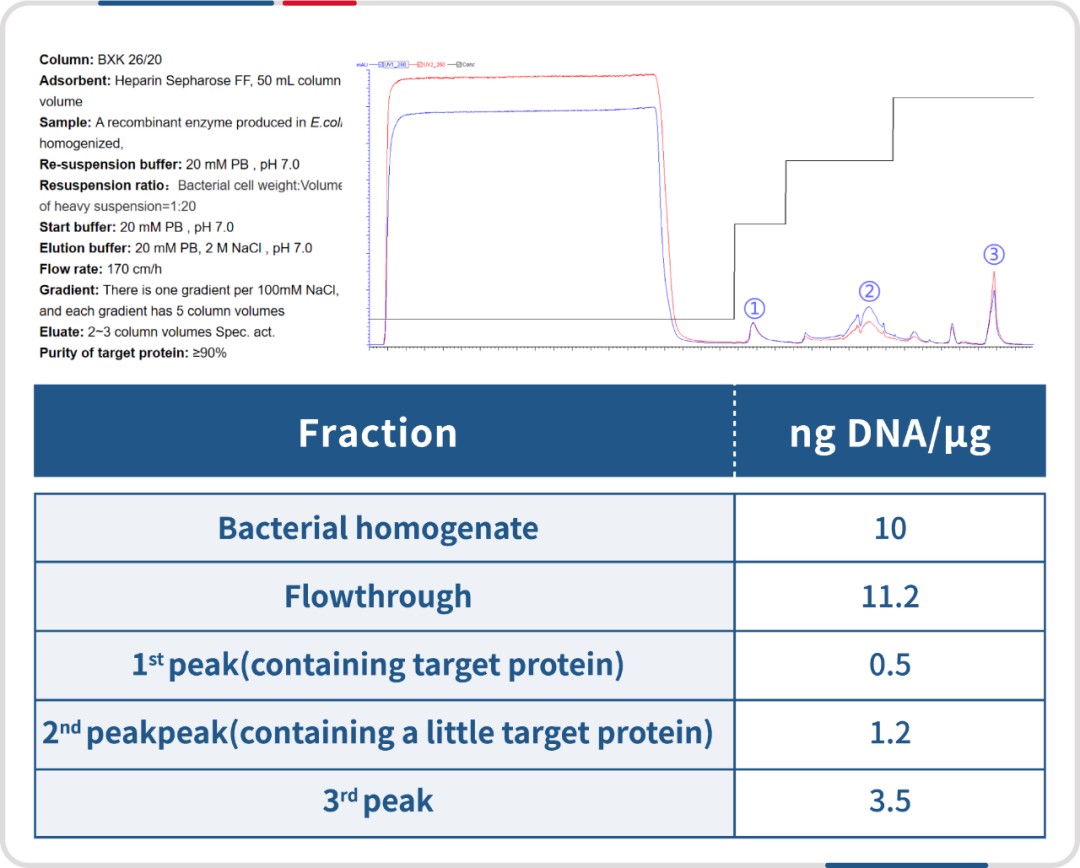
图注:通过肝素层析,HCD 可以从 10 ng/μg 降低到 0.5 ng/μg
→阴离子交换层析
核酸可以与阴离子交换层析填料较紧密地结合,通过设置不同的洗脱梯度,能有效地分离目的蛋白与核酸杂质;Q FF 层析用于某酵母表达的蛋白酶捕获,结果显示洗脱峰[1] 对应的目标蛋白中 HCD 含量相比粗品降低了大约 10 倍。
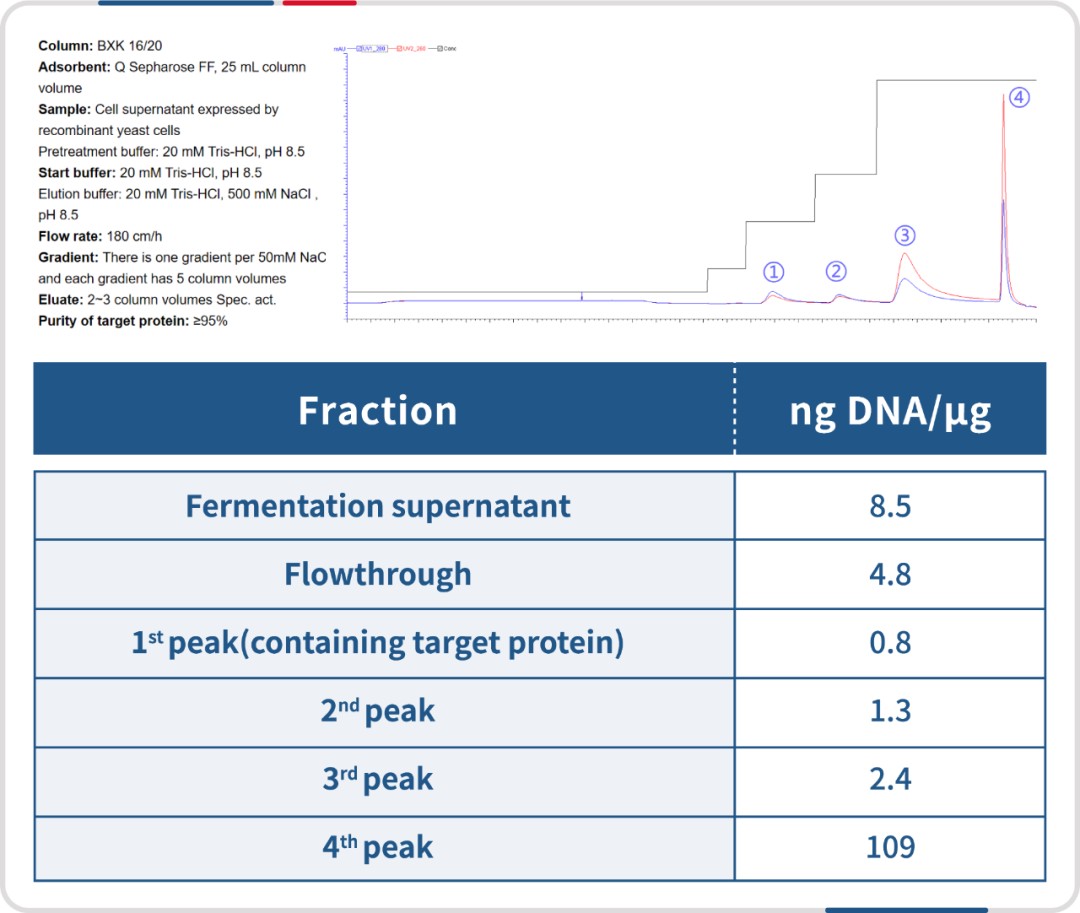
图注:通过阴离子层析,HCD 可以从 8.5 ng/μg 降低到 0.8 ng/μg
→凝胶过滤层析采用凝胶层析分离核酸往往伴随着对样品的预处理,使得核酸与蛋白提前解离。在 CREB 转录因子的纯化中,尝试变性、高盐的条件下进行凝胶过滤层析,纯化后再对目的蛋白进行复性,最终有效地改善了目的蛋白的核酸污染问题[10],当然,超滤分离模式也能起到类似效果。
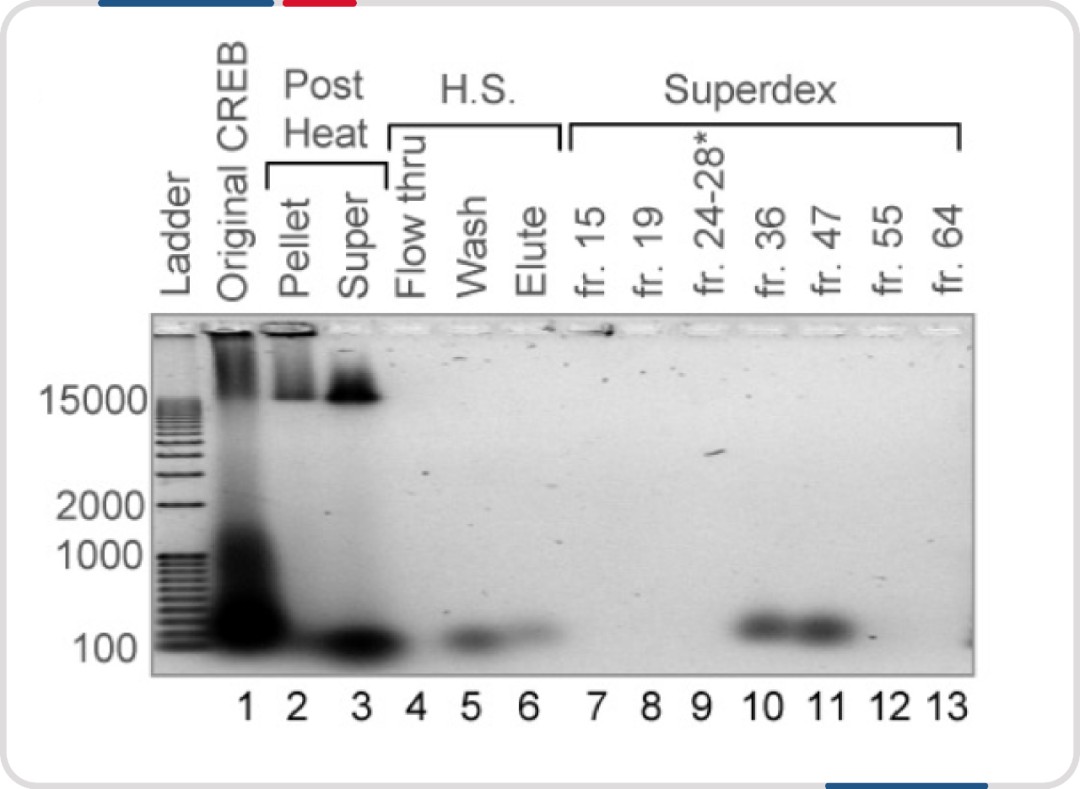
图注:CREB 粗品(泳道 1)经过变性后,通过 Superdex 凝胶层析有效分离了目标蛋白(泳道 9)和核酸污染物(泳道 5,6,10 和11)
→疏水和复合层析核酸含有大量的含氧基团,能够与水分子形成氢键,从而具有良好的亲水性。相反,某些蛋白质中富含亮氨酸、异亮氨酸和缬氨酸等氨基酸,疏水性则会较强,对于这类蛋白可以利用疏水层析分离去除核酸。越来越多的复合层析介质如 Capto Adhere、CHT 等也在生物大分子的核酸去除中发挥着重要的作用。Konstantinidis 等[11] 将澄清后麻疹病毒粗品分别用 Capto 700 和 Capto Adhere 进行纯化,在同滴度下 Capto Adhere 的 HCD 残留最低。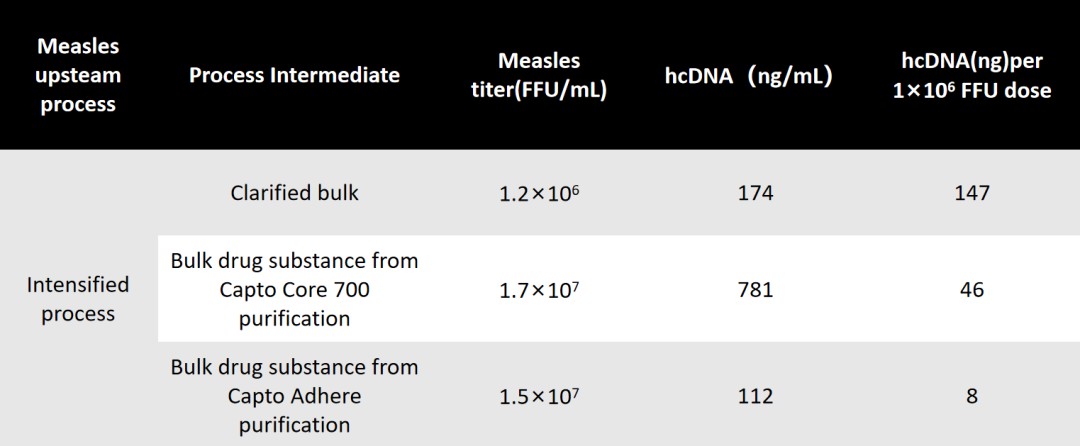
图注:上游两种上清分别进行了 Capto 700 及 Capto Adhere 的纯化对比,Capto Adhere 对 HCD 的去除更有效
→层析优化和组合小技巧合理的层析条件设置能够让分离的优势发挥到极致,对于较难去除的核酸残留,还可以尝试进行切峰收集,在大肠表达的重组 M-MLV 逆转录酶用亲和层析分离过程,对洗脱组分的峰前、峰中、峰后(30%
UVmax~ UVmax ~ 30% UVmax )进行核酸残留量分析,发现核酸残留的分布有较大的差异。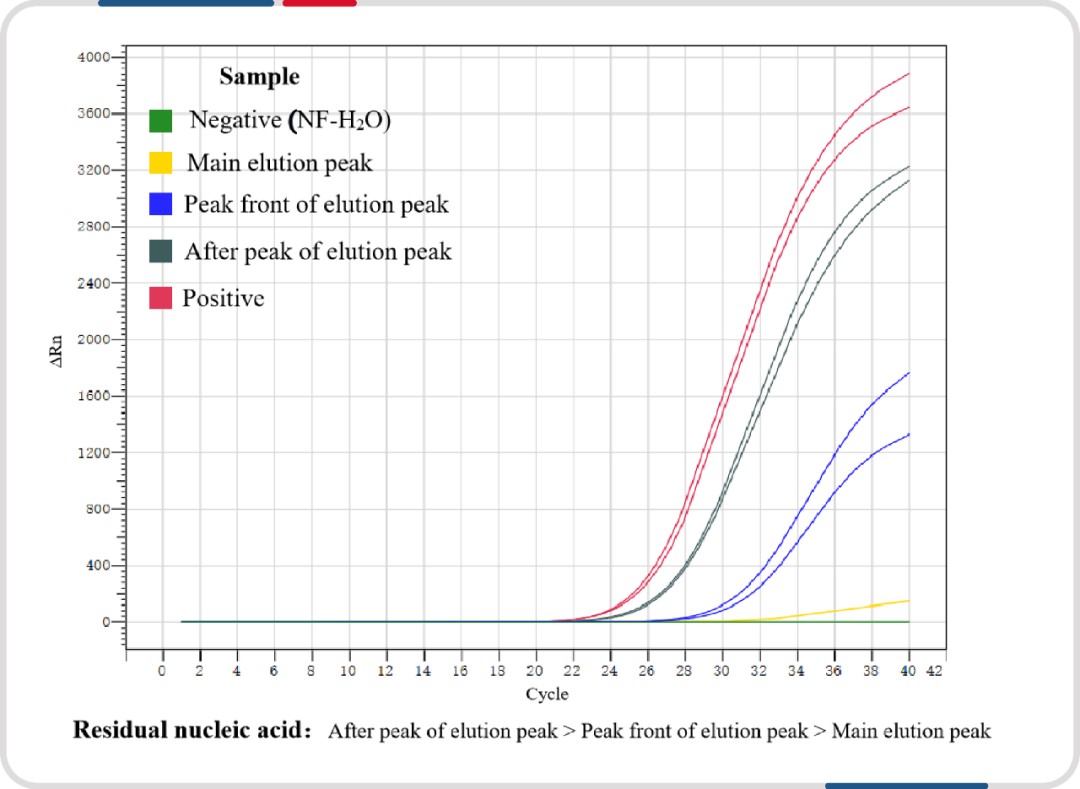
图注:通过切峰收集,峰后组分的核酸残留量>主峰>峰前
层析组合的先后顺序也对核酸残留去除效果不同。在 T4 DNA 连接酶开发时,筛选获得了疏水、亲和和阴离子这三个优势填料,随后进行了不同顺序组合,其中亲和→疏水→阴离子层析(Seq.3)的 HCD 残留是
0.000015pg/μL,疏水→亲和→阴离子(Seq.1)的 HCD 残留是 0.2pg/μL,Seq.3 的 HCD 去除效果是
Seq.1 的上万倍。
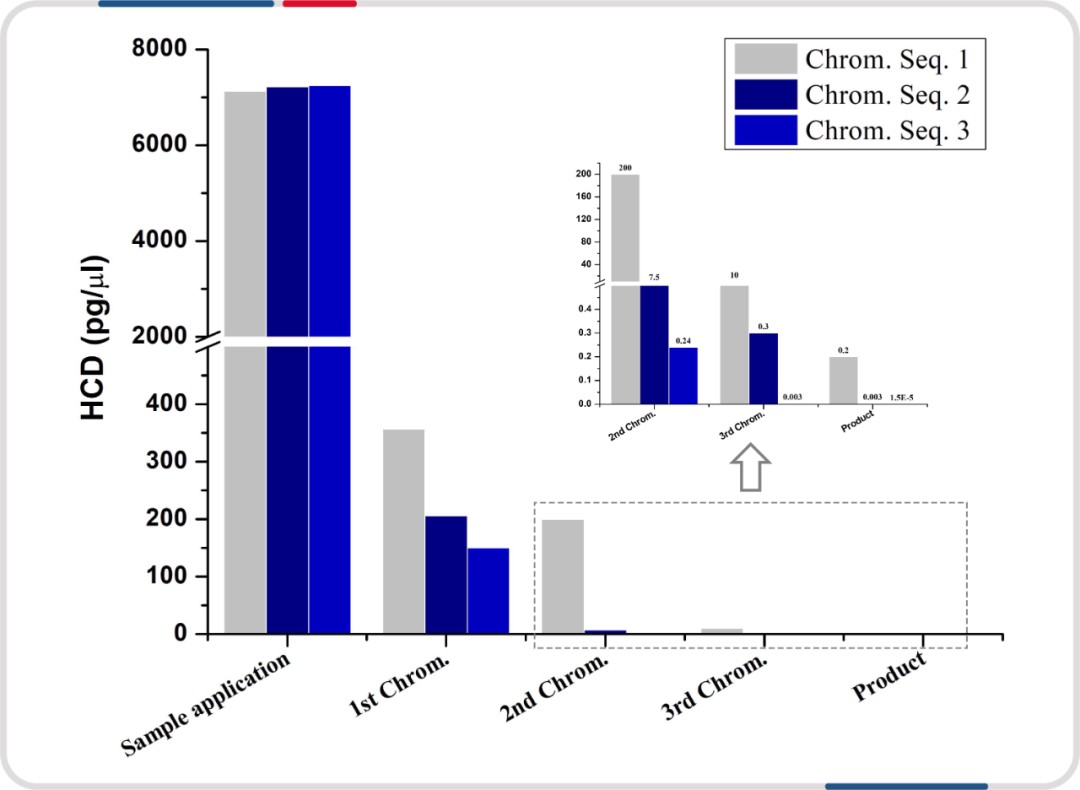
图注:进行亲和、疏水和阴离子不同顺序组合,其中疏水→亲和→阴离子(Seq.1)的成品 HCD 为 0.2pg/μL,阴离子→亲和→疏水(Seq.2)工艺的成品 HCD 为
0.003pg/μL,亲和→疏水→阴离子工艺(Seq.3)的成品 HCD 为 0.000015pg/μL。
核酸残留水平往往只是工艺开发的目标之一,在工艺优化和组合的过程中我们需要进行整体工艺目标的跟踪和把控,Silva R M 等[12] 对纯化工艺不同阶段的目标物回收率、核酸残留和蛋白残留等进行记录,通过衡量各维度数据,进行最佳工艺的确认。
03 超净之选!瀚海新酶高洁净分子酶和开发平台
高洁净的分子酶
经过多年的沉淀和积累,瀚海新酶建立了完善的超低宿主细胞核酸残留的研发和生产平台,通过严格的质量体系控制,完成了高质量的分子酶系列的开发,如 Taq DNA 聚合酶酶系列、M-MLV 逆转录酶系列和 mNGS 蛋白酶 K 系列等,这些酶都具有极低的核酸残留。
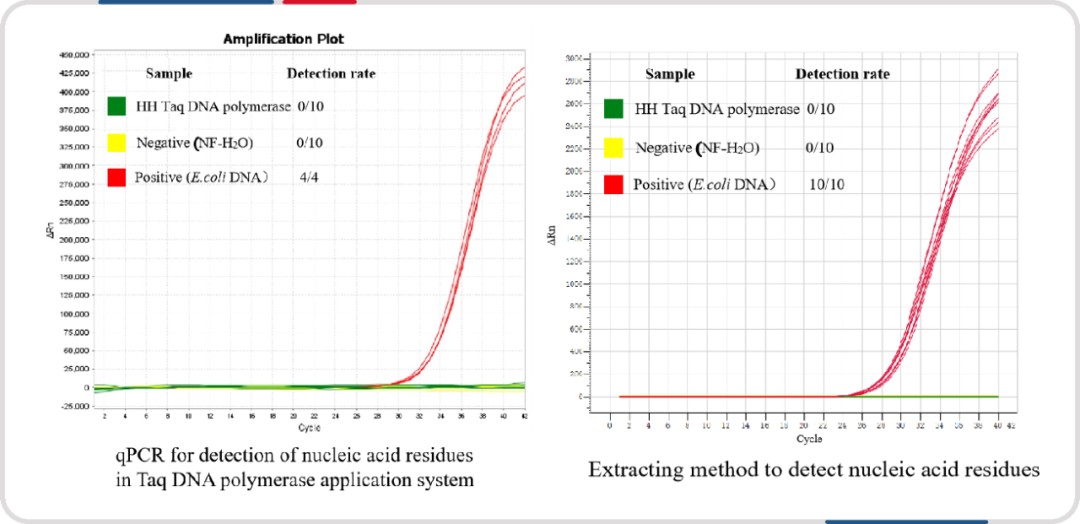
图注:不管是采用针对性强的应用端检测方法,还是普适性强的试剂盒抽提法,瀚海新酶的Taq DNA polymerase 的核酸残留均是无检出。
订 购 信 息
重组蛋白CMO平台
瀚海新酶作为特种酶行业领先者拥有丰富的重组蛋白产品开发和生产经验,,经过长期的项目研发、核心技术积累以及产业化深耕,我们搭建了超净酶的纯化平台和与之匹配的厂房设备设施,可匹配小试-中试-产业化各个阶段的生产需求。

灵活的产能匹配
参考文献
[1.] Huang Y, Sun L, Liu W, et al. Multiplex single-cell droplet PCR with machine learning for detection of high-risk human papillomaviruses[J]. Analytica Chimica Acta, 2023, 1252: 341050.
[2.] Nadiya S, Kolla H B, Reddy P N. Optimization and evaluation of a multiplex PCR assay for detection of Staphylococcus aureus and its major virulence genes for assessing food safety[J]. Brazilian Journal of Microbiology, 2023, 54(1): 311-321.
[3.] Kim S H, Lee S Y, Kim U, et al. Diverse methods of reducing and confirming false-positive results of loop-mediated isothermal amplification assays: A review[J]. Analytica Chimica Acta, 2023: 341693.
[4.] Zhang C, Li Z, Liu J, et al. Synthetic gene circuit-based assay with multilevel switch enables background-free and absolute quantification of circulating tumor DNA[J]. Research, 2023, 6: 0217.
[5.] 李云龙, 张健, 魏艳秋, 等. 分子诊断实验室去除核酸污染的方法学研究[J]. 生物工程学报, 2021, 37(2): 673-679.
[6.] Zhang S M, Roberts M, Jones M, et al. A novel method for removing polyethyleneimine from biopharmaceutical samples: improving assay sensitivity of residual DNA qPCR[J]. BioTechniques, 2020, 68(6): 353-358.
[7.] Li S M, Bai F L, Xu W J, et al. Removing residual DNA from Vero-cell culture-derived human rabies vaccine by using nuclease[J]. Biologicals, 2014, 42(5): 271-276.
[8.] Kawka K, Wilton A N, Madadkar P, et al. Integrated development of enzymatic DNA digestion and membrane chromatography processes for the purification of therapeutic adenoviruses[J]. Separation and Purification Technology, 2021, 254: 117503.
[9.] Radosevich M, Burnouf T. Affinity chromatography–fractionated and DNA-engineered plasma proteins[J]. Encyclopedia of Industrial Biotechnology: Bioprocess, Bioseparation, and Cell Technology, 2009: 1-12.
[10.] Dinaida I. Lopez, et al. Purification of CREB to apparent homogeneity: removal of truncation products and contaminating nucleic acid. Protein Expr Purif. 55, 406-418 (2007).
[11.] Konstantinidis S, Poplyk M R, Ma W, et al. Purification processes of live virus vaccine candidates expressed in adherent Vero cell lines via multimodal chromatography in flowthrough mode[J]. Biotechnology and Bioengineering, 2024, 121(8): 2482-2499.
[12.] Silva R M, Rosa S S, Cunha R, et al. Anion exchange chromatography-based platform for the scalable purification of extracellular vesicles derived from human mesenchymal stromal cells[J]. Separation and Purification Technology, 2023, 310: 123238.





 USA
USA








